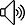如下所示:
#-*- coding: utf-8 -*-
import pandas as pd
import numpy as np
from patsy.highlevel import dmatrices
#2.7里面是from patsy import dmatrices
from statsmodels.stats.outliers_influence import variance_inflation_factor
import statsmodels.api as sm
import scipy.stats as stats
from sklearn.metrics import mean_squared_error
import seaborn as sns
import matplotlib.pyplot as plt
import matplotlib.mlab as mlab
import matplotlib
#數據獲取
ccpp = pd.read_excel('CCPP.xlsx')
ccpp.describe()
#繪制各變量之間的散點圖
sns.pairplot(ccpp)
plt.show()
#發(fā)電量(PE)與自變量之間的相關系數
a = ccpp.corrwith(ccpp.PE)
print(a)
#將因變量PE,自變量AT,V,AP和截距項(值為1的1維數值)以數據框的形式組合起來
y,x = dmatrices('PE~AT+V+AP',data = ccpp,return_type = 'dataframe')
#構造空的數據框
vif = pd.DataFrame()
vif[""VIF Factor""] = [variance_inflation_factor(x.values,i) for i in range(x.shape[1])]
vif[""features""] = x.columns
print (vif)
#構建PE與AT,V和AP之間的線性模型
fit = sm.formula.ols('PE~AT+V+AP',data=ccpp).fit()
b = fit.summary()
# print(b)
#計算模型的RMSE值
pred = fit.predict()
c = np.sqrt(mean_squared_error(ccpp.PE,pred))
print(c)
#離群點檢驗
outliers = fit.get_influence()
#高杠桿值點(帽子矩陣)
leverage = outliers.hat_matrix_diag
#dffits值
dffits = outliers.dffits[0]
#學生化殘差
resid_stu = outliers.resid_studentized_external
#cook距離
cook = outliers.cooks_distance[0]
#covratio值
covratio = outliers.cov_ratio
#將上面的幾種異常值檢驗統(tǒng)計量與原始數據集合并
contat1 = pd.concat([pd.Series(leverage,name = 'leverage'),pd.Series(dffits,name ='dffits'),
pd.Series(resid_stu,name = 'resid_stu'),pd.Series(cook,name = 'cook'),
pd.Series(covratio,name ='covratio'),],axis = 1)
ccpp_outliers = pd.concat([ccpp,contat1],axis = 1)
d = ccpp_outliers.head()
print(d)
#計算異常值數量的比例
outliers_ratio = sum(np.where((np.abs(ccpp_outliers.resid_stu)>2),1,0))/ccpp_outliers.shape[0]
e = outliers_ratio
print(e)
#刪除異常值
ccpp_outliers = ccpp_outliers.loc[np.abs(ccpp_outliers.resid_stu)<=2,]
#重新建模
fit2 = sm.formula.ols('PE~AT+V+AP',data = ccpp_outliers).fit()
f = fit2.summary()
# print(f)
pred2 = fit2.predict()
g = np.sqrt(mean_squared_error(ccpp_outliers.PE,pred2))
print(g)
#
#殘差的正態(tài)性檢驗(直方圖法)
resid = fit2.resid
#中文和負號的正常顯示
# plt.rcParams['font.sans=serif'] = ['Microsoft YaHei']
plt.rcParams['font.sans-serif'] = ['SimHei']
# plt.rcParams['font.sans=serif'] = 'sans-serif'
plt.rcParams['axes.unicode_minus'] = False
plt.hist(resid,bins = 100,normed = True,color = 'steelblue',edgecolor = 'k')
#設置坐標軸標簽和標題
plt.title('殘差直方圖')
plt.ylabel('密度值')
#生成正態(tài)曲線的數據
x1 = np.linspace(resid.min(),resid.max(),1000)
normal = mlab.normpdf(x1,resid.mean(),resid.std())
#繪制正態(tài)分布曲線
plt.plot(x1,normal,'r-',linewidth = 2,label = '正態(tài)分布曲線')
#生成核密度曲線的數據
kde = mlab.GaussianKDE(resid)
x2 = np.linspace(resid.min(),resid.max(),1000)
#繪制核密度曲線
plt.plot(x2,kde(x2),'k-',linewidth = 2,label = '核密度曲線')
#去除圖形頂部邊界和右邊界的刻度
plt.tick_params(top = 'off',right = 'off')
#顯示圖例
plt.legend(loc='best')
#顯示圖形
plt.show()
#生成的正態(tài)曲線的數據
pp_qq_plot = sm.ProbPlot(resid)
pp_qq_plot.ppplot(line = '45')
plt.title('P-P圖')
pp_qq_plot.qqplot(line = 'q')
plt.title('Q-Q圖')
plt.show()
#殘差的正態(tài)性檢驗(非參數法)
standard_resid = (resid-np.mean(resid))/np.std(resid)
g = stats.kstest(standard_resid,'norm')
print(g)
# 總結:由于shapiro正態(tài)性檢驗對樣本量的需求是5000以內,而本次數據集樣本量有9000多,故選擇k-s來完成正態(tài)性檢驗。
# 從k-s檢驗的p值來看,拒絕了殘差服從正態(tài)分布的假設,即認為殘差并不滿足正態(tài)性假設這個前提。
# 如果殘差不服從正態(tài)分布的話,建議對Y變量進行box-cox變換處理。
# 由于fit2模型的殘差并沒有特別明顯的偏態(tài)(偏度為0.058,接近于0),故這里就不對Y進行變換。
#
# import scipy.stats as stats
# #找到box-cox變換的Lambda系數
# lamd = stats.boxcox_normmax(vif.y,method = 'mle')
# #對y進行變換
# vif['trans_y'] = stats.boxcox(vif.y,lamd)
# #建模
# fit3 = sm.formula.ols('y~x1+x2...',data = vif).fit()
# fit3.summary()
以上這篇python3 線性回歸驗證方法就是小編分享給大家的全部內容了,希望能給大家一個參考,也希望大家多多支持腳本之家。
更多文章、技術交流、商務合作、聯(lián)系博主
微信掃碼或搜索:z360901061

微信掃一掃加我為好友
QQ號聯(lián)系: 360901061
您的支持是博主寫作最大的動力,如果您喜歡我的文章,感覺我的文章對您有幫助,請用微信掃描下面二維碼支持博主2元、5元、10元、20元等您想捐的金額吧,狠狠點擊下面給點支持吧,站長非常感激您!手機微信長按不能支付解決辦法:請將微信支付二維碼保存到相冊,切換到微信,然后點擊微信右上角掃一掃功能,選擇支付二維碼完成支付。
【本文對您有幫助就好】元